近期,納米生物醫學領域知名學術期刊《納米生物技術雜誌》(Journal of Nanobiotechnology) 發表了万博英超狼队网官方网 、遺傳工程國家重點實驗室黃強教授課題組關於抗新冠病毒 (SARS-CoV-2) 蛋白藥物設計的研究論文:Computational design and engineering of self-assembling multivalent microproteins with therapeutic potential against SARS-CoV-2。該論文旨在設計自組裝型多價蛋白束,通過多位點協同結合增強其與新冠病毒變異株的結合親和力,實現廣譜中和多種變異病毒株,為應對病毒變異提供新策略。
新冠疫情的高峰期雖已過去,但新冠病毒還在持續變異,產生能逃避免疫係統的新變種,使現有疫苗和抗病毒藥物,特別是中和抗體蛋白藥物失效。抗病毒蛋白藥物通常基於人體在病毒感染或疫苗免疫時產生的天然抗體開發而成,它們主要識別並結合病毒表麵的刺突(簡稱S) 蛋白的受體結合域 (簡稱RBD),阻止病毒與人宿主細胞的ACE2受體結合,進而抑製病毒入侵和複製。然而,變異病毒RBD區域的氨基酸突變削弱了蛋白藥物的結合能力,使其無法有效阻斷病毒與宿主細胞的結合。例如,Omicron變異株的突變使得許多早期抗體藥物失效。因此,對天然抗體蛋白進行改良,人工設計出能夠中和多種變異病毒的廣譜性蛋白藥物顯得尤為重要。
黃強團隊的論文根據S蛋白為同源三聚體的結構特點,提出了一種創新的蛋白藥物設計策略,核心是通過多個納米抗體或微型蛋白的有序自組裝,設計能同時結合到S蛋白三個RBD上的多價蛋白束,如多價納米抗體束 (Multivalent nanobody cluster)。利用多位點協同結合產生的相互作用累加效應,彌補單個位點上因氨基酸突變而減弱的親和力,從而顯著提升蛋白藥物與變異病毒的親和力,使之能夠中和多種病毒變異株,實現廣譜治療的目的。
與以往研究常采用的經驗試錯法不同,本研究首先開發了一套基於物理模型的計算設計方法,用於自組裝多價蛋白。這套方法旨在設計出自組裝效率高、構象均一性好的多價體,為後續的蛋白質藥物開發奠定堅實的基礎。該方法分為4步:基於結構的多價體連接子設計、多價體的分子模擬評估、多價體自組裝狀態的實驗驗證,以及多價蛋白的功能測試 (圖1)。以微型蛋白 (簡稱為MP) miniACE2為RBD的單價結合劑,對多價體三聚化支架、支架連接子類型和長度等因素進行了設計,成功獲得了一個自組裝效率高、構象均一好的三價微蛋白束MP-5ff。分子互作實驗顯示,與親和力原本就較高的單價結合劑相比,MP-5ff的RBD親和力提升了約1000倍,平衡解離常數 (Kd) 達到小於1 pM的水平。
圖1:自組裝型多價微蛋白束的計算設計與實驗驗證流程。
在此基礎上,以納米抗體Nb67為RBD的單價結合劑,應用MP-5ff的三聚化支架和連接子,構建了自組裝特性同樣優良的三價納米抗體束Tr67 (圖2)。Tr67展現出了比Nb67更強的結合親和力,具有廣譜的抗病毒活性。假病毒中和實驗表明,Tr67能夠有效抑製Omicron病毒的主要變異株,包括納米抗體Nb67無法中和的變異株,以及對現有抗體逃逸性最強的XBB.1和XBB.1.5變異株。

圖2:三價納米抗體束Tr67的設計和表征。
最後,冷凍電鏡結構分析確認,Tr67確實以三價模式與Omicron的S蛋白結合,並且結合在該蛋白較為罕見的“3-RBD-up”活性構象上 (圖3)。這一發現表明,Tr67能夠抑製病毒表麵那些可與ACE2受體結合的活性構象。有研究表明,把S蛋白鎖定在“3-RBD-up”構象還有助於免疫係統識別和清除病毒。
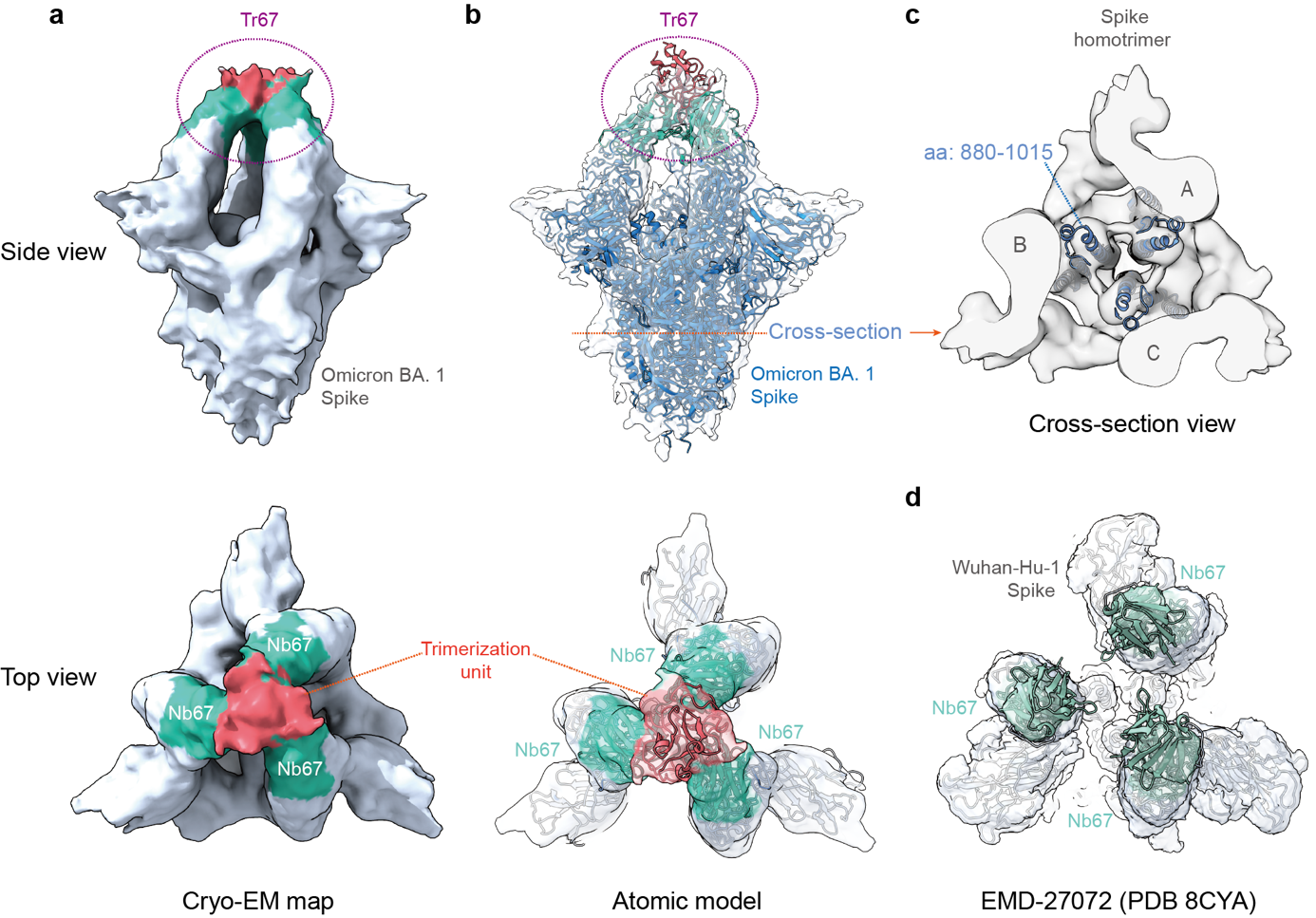
圖 3. 納米抗體束Tr67結合Omicron病毒S蛋白的“3-RBD-up”構象。
在許多情況下,使用傳統抗體篩選技術尋找既具有高親和力又擁有強廣譜中和能力的天然抗體往往非常困難,有時甚至無法實現目標。而本研究最重要的啟示在於:采用多價化設計方法,改造那些親和力和廣譜性一般但相對容易獲得的抗體,可以顯著提升其親和力和廣譜中和能力。這不僅為迅速開發抗新冠變異病毒的治療藥物提供了新的途徑,而且也適用於針對其他致病性病毒的蛋白藥物研發。
万博英超狼队网官方网 碩士研究生秦琴同學為論文的第一作者,黃強教授為通訊作者,薑欣怡同學等其他作者參與了研究工作。論文研究獲得了國家自然科學基金和國家科技重大專項項目、遺傳工程國家重點實驗室等的支持。
論文鏈接:https://doi.org/10.1186/s12951-024-02329-3 (J. Nanobiotech. 2024, 22:58)

